病毒驱动型和致癌物驱动突变型头颈癌的免疫状况
5.在HPV + TIL中发现了生发中心B细胞
已经发现TME中的B细胞与人类肿瘤类型的总体生存呈正相关,并会影响CD4 + Tconv细胞表型。B细胞聚类显示11个亚群(图4A),基因集富集分析确定了:生发中心B细胞(#1-4)、浆细胞(#5)、幼稚细胞(#6和9),记忆细胞(#8和11)(图4C)。细胞周期富集提示#3和4为暗区中迅速增殖的B细胞,而#1和2为亮区的中心细胞,在此处它们会受到CD4 + TFH的选择。而在HPV–TIL中,仅存在于血浆或早期的B细胞(图4B)可能与缺乏CD4 + TFH有关(图3BE)。B细胞的扩散映射图(图4DE)显示了跨多轴分化的复杂过程,可以看到DC1与生发中心形成有关,DC4与从幼稚到记忆B细胞的过渡有关以及DC3与向浆细胞的发育相关,其中HPV–TIL和HPV+TIL在DC1上有显著的差异,提示生发中心B细胞的显著差异。生发中心B细胞存在于HPV+ HNSCC各个发展阶段,而HPV–HNSCC中心B细胞较少,提示HPV + HNSCC可能有更好的预后。


图4. B细胞分析揭示了生发中心B细胞和相关的独特B细胞群体
6.共同的轨迹产生不同的髓系状态谱
髓样细胞有肿瘤相关巨噬细胞(TAMs)和树突状细胞。TAM可以抗肿瘤的M1样或促肿瘤的M2样。最近研究表明TME中髓样细胞之间存在更广泛的异质性,或可共同表达或独特表达。这里确定了8个髓样细胞亚群,其中#2-4主要存在于PBMC中,而#1和#5-8主要存在于TIL中(图5AB)。
#2为CD16 +单核细胞,#3和4为CD14 +单核细胞(CD16+更成熟),且#3的FCGR3A(CD16)表达较低,表明它不是CD14 + CD16 +单核细胞的过渡亚群。#5和7则表达高水平的趋化因子和细胞因子。#1、6和8表达较高的HLA分子(与抗原呈递细胞一致),但#1的IDO1,CCL17和CCR7表达较高;#6表达CD1C(与常规树突状细胞一致);#8高表达补体相关基因,以及M2相关基因。在HPV–TILs中发现有更多的#1和8,提示HPV–疾病患者具有潜在的免疫抑制作用。
接着进行扩散图映射,并确定了几个分支(图5DE),#5位于中心,在DC1和DC2轴上的正方向上,#5朝#7和8发展,提示#7和8为终末分支(图5EG)。已知补体高表达与未成熟的树突状细胞相关,表明促进#8这个树突状细胞亚群的成熟可以改善抗肿瘤T细胞反应。(图5G)

图5. 髓系细胞在TME中的独特状态和潜在的可塑性
7.免疫细胞之间的crosstalk
单细胞转录组学分析不仅可以揭示细胞内在信息,还可以通过配体和受体表达来探究可能的细胞外相互作用。为了找到潜在的细胞间相互作用,使用了708种配体和691种受体的list来鉴定配体/受体的表达,这些配体/受体可以形成总共2557个潜在的相互作用对。首先评估了HPV–和HPV + HNSCC中差异表达的配体和受体(图6AB)。接下来,为了挖掘更大的潜在配体和受体作用,使用自主开发的R包CellTalker(https://github.com/arc85/celltalker)鉴定免疫谱系内部和谱系之间的相互作用。总共鉴定了168个配体和194个受体,它们参与了481个独特的相互作用,最后用circos图进行可视化(图6C-6F)。交互大致分为三种类型:common、unique(与健康供体相比)、unique(与所有其他样本相比)。可以看到,与健康人的PBMC和tonsil相比,TIL中的细胞通讯更多。


图6.HPV–和HPV + TIL之间的受体和配体表达模式不同,并且在TIL中发生广泛的细胞间通讯
8.空间组织与转录特征一致
尽管上一部分的分析从全局的角度揭示了细胞交互,但了解免疫细胞的空间定位将为基于邻近性推测的细胞间通讯提供更严格的论证。所以接下来对同一患者的成对组织切片进行了多光谱免疫荧光(IF)来探索免疫和肿瘤细胞的空间位置,并基于邻近性确定了可能参与细胞间通讯的免疫亚群。
首先根据每种免疫细胞在亚群的频率对每个感兴趣区域(ROI)进行分层聚类(图7A),揭示了与特定免疫谱系相关的聚类。每张图像中,使用R包trimesh进行了Delunay三角剖分(Delunay triangulation,一种图像处理技术),基于邻近度来识别最可能彼此交互细胞类型。图7B-7F右图为一种细胞类型与另一种类型相互作用的对数比几率。在B细胞频率最高的#3和5中,B细胞相互作用几率最高的是其他B细胞,其次是CD4 + Tconv(图7D和7F)。基于空间关系,接下来使用CellTalker来推断HPV–和HPV + TIL中B细胞和CD4 + Tconv细胞之间交互网络的差异(图7G和7H)。与scRNA-seq和IF分析一致,发现HPV–和HPV + TILs中均有B细胞和CD4 + Tconv细胞的相互作用,但只有HPV + TIL的生发中心B细胞(cluster B-1至cluster B-4)与TFH(clusterCD4+ -1)之间有相互作用。
接下来,利用来自TCGA的临床数据和bulk mRNA数据确定HNSCC中的CD4 + TFH是否与生存相关。与之前发表的数据,HPV +患者的预后比HPV -好。然后,根据来自scRNA-seq数据中与TFH相关的差异表达基因评估了患者的TFH基因signature,并根据TFH评分进行分层以进行单变量PFS分析(图7I)以及多变量生存分析,高TFH均与较好的预后显著相关。总的来说,确定了不同组织切片的独特免疫浸润模式,并使用CellTalker以及HPV +和HPV-HNSCC中的scRNA-seq数据表征了独特的TFH亚群和生发中心的相互作用,并发现TFH富集的转录特征与更好的预后相关。

图7. IF分析提供了基于空间定位的推定细胞间通讯信息
小结
本篇学习笔记利用scRNA测序评估了HPV–和HPV + HNSCC患者以及健康供体的外周和肿瘤内免疫群体中131,224个单细胞的转录谱,转录组结果通过多光谱免疫荧光分析和基于空间邻近性来推测细胞交互来进行背景分析。发现了HPV–和HPV + HNSCC免疫谱系之间的差异,这些结果对免疫疗法的设计将产生重要的影响,相似的免疫谱系(CD8 + T之间和CD4 + Treg之间)靶向疗法可能差异不大,而针对不同的免疫谱系(即CD4 + Tconv细胞,B细胞和髓样细胞)的免疫疗法则较为多变。
这篇学习笔记有很多亮点,首先新开发的三种算法:DRAGON:基于热学知识进行聚类、SingleSetGset进行基因集富集测试、CellTalker用于推断细胞交互;其次是研究主题更有转化意义,一般来说很多都是研究不同分子分型之间的差异,而这篇学习笔记着眼于病因差异研究,更容易在临床上进行判断;然后研究内容比较丰富,不仅基于单细胞测序信息分析了特定免疫谱系的亚群状态,还分析了细胞间通讯和细胞邻近性关系。最后是研究中的统计学分析也很严谨,不仅在鉴定差异基因用了事后功效分析,在细胞聚类也用其进行统计学验证。在比较免疫谱系差异时,也较新颖地使用了巴氏距离来量化差异。

图片新闻
技术文库
最新活动更多
-
7.30-8.1火热报名中>> 全数会2025(第六届)机器人及智能工厂展
-
7月30-31日报名参会>>> 全数会2025中国激光产业高质量发展峰会
-
精彩回顾立即查看>> 【线下论坛】新唐科技2025新品发布会
-
精彩回顾立即查看>> 【在线会议】研华嵌入式核心优势,以Edge AI驱动机器视觉升级
-
精彩回顾立即查看>> OFweek 2025(第十四届)中国机器人产业大会
-
精彩回顾立即查看>> 【在线会议】从直流到高频,材料电特性参数的全面表征与测量
-
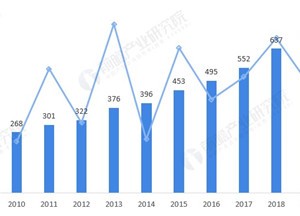
10 “意外”的药明康德







 分享
分享










发表评论
请输入评论内容...
请输入评论/评论长度6~500个字
暂无评论
暂无评论